Wie neue Ansätze beim Finden und Analysieren großer Datenmengen helfen
von Prof. Dr. Konrad Förstner
In der Mikrobiologie besteht ein großes Ungleichgewicht bei den Informationen über verschiedene Arten von Bakterien. Für die große Mehrheit der Bakterien sind bestenfalls Sequenzinformationen aus dem Genom verfügbar. Das Gemeinschaftsprojekt DiASPora von ZB MED, DSMZ und TIB wendet KI-basierte Ansätze an, um die Lücke zu schließen. Ausgangspunkt für diesen Ansatz ist die Datenbank BacDive, die größte Quelle für standardisierte phänotypische Daten über Bakterien. Im Projekt DiASPora wird die Datenbasis durch Text-Mining-Verfahren erweitert, um phänotypische Daten aus der Literatur zu extrahieren und genomische Phänotypen zu ermitteln. Dies bildet den Ausgangspunkt für eine KI-gestützte Vorhersage der Physiologie und der geeigneten Kultivierungsbedingungen für noch nicht kultivierte Bakterien.
Bakterien lassen sich grob in drei Gruppen einteilen:
- Einige wenige gut untersuchte Modellorganismen.
- Tausende von Arten, die kultiviert werden konnten, aber noch nicht im Detail untersucht wurden.
- Vermutlich Millionen von Organismen, die bestenfalls in Sequenzierungsdatensätzen vorkommen und bisher weder kultiviert noch untersucht wurden.
DiASPora befasst sich mit den beiden letztgenannten Fällen, um Licht in das Dunkel rund um die wenig erforschten Bakterien zu bringen. Und dabei kommt Künstliche Intelligenz zum Einsatz. Mit Hilfe von Natural-Language-Processing-Ansätzen (NLP) werden vorhandene Informationen aus publizierten Fachartikeln extrahiert. DiASPora stützt sich dabei auf die umfassenden Datensätze von BacDive.

BacDive
Die Bacterial Diversity Metadatabase ist die weltweit größte Datenbank für standardisierte phänotypische Informationen von Bakterien. BacDive umfasst Daten zu über 19.000 Arten und über 90.000 Stämmen. Die Daten decken über 600 verschiedene Datenfelder ab, darunter Taxonomie, Morphologie, Physiologie und Kultivierungsbedingungen. Aufgrund der standardisierten Datenfelder ermöglicht BacDive systematische Analysen, wie zum Beispiel Vergleiche über eine Vielzahl von Bakterienarten, sowie das Auffinden von Stämmen anhand bestimmter Merkmale. Der Umfang der Datenbank weist jedoch noch Lücken auf, die geschlossen werden müssen, um umfassende Analysen über alle bekannten Arten zu ermöglichen.
BacDive ist ein Angebot vom Leibniz-Institut DSMZ-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH in Braunschweig. Die Datenbank entspricht den FAIR-Prinzipien und ist frei verfügbar.
Wo sich Daten verstecken
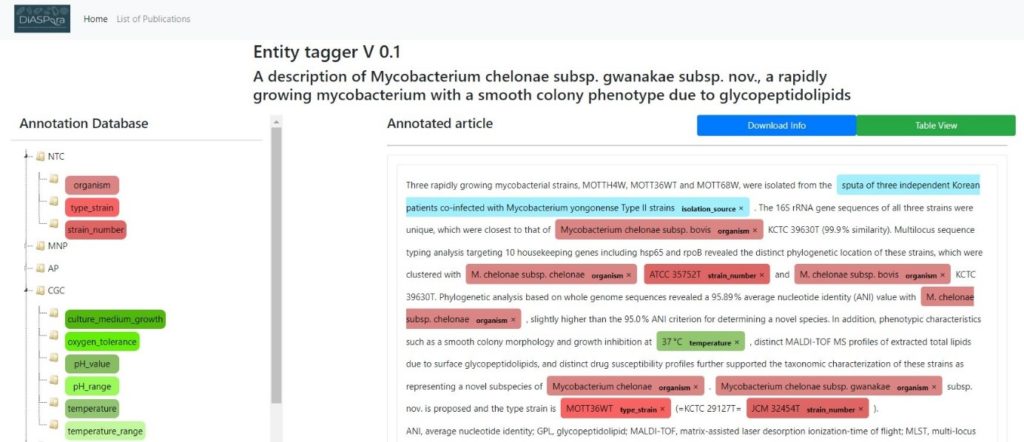
Die Beschreibung von Organismen ist seit jeher ein grundlegender und wesentlicher Bestandteil für die Untersuchung der Vielfalt, Taxonomie und Evolution in den Biowissenschaften. Wissenschaftler:innen haben im Laufe der Jahrhunderte auf diese Weise riesige Mengen an Literatur angehäuft, die umfassende taxonomische und phänotypische Informationen für jede in ihren Forschungsarbeiten behandelte Art liefert. Die Datenbank BacDive, die von der DSMZ betrieben wird, bietet bereits Zugang zu standardisierten Daten für eine Vielzahl an Artenbeschreibungen von Bakterien. Bisher wurden die Informationen mit erheblichem Aufwand in einem manuellen Workflow aus der Literatur entnommen: Auffinden der Informationen, Extraktion der relevanten Kriterien und schließlich die Umwandlung in standardisierte BacDive-Datensätze. Auch wenn es sich um Informationen handelt, die öffentlich verfügbar sind, verstecken sich die Daten in unstrukturiertem Text. Zudem ist die natürliche Sprache sehr variabel. Um diese versteckten Daten aufzuspüren – und damit der Forschung zugänglich zu machen – wendet das DiASPora-Team eine Kombination aus regelbasierten und KI-basierten Modellen zur Informationsextraktion an. Dabei stellen sie Beziehungen zwischen verschiedenen Entitäten und anderen Satzteilen her.
Damit eine Anwendung auf Basis von maschinellen Lernverfahren zuverlässig arbeiten kann und qualitativ hochwertige Ergebnisse liefert, braucht sie robuste Trainingsdaten. Um die zu erhalten, ist es notwendig, mehrere Verfahren – automatische und manuelle – parallel und ineinander greifend anzuwenden. Ziel all dieser vorbereitenden Arbeiten ist es, einen automatisierten Ansatz zu entwickeln, der dann durch Feedback-Zyklen mit Expert:innen der jeweiligen Domänen die robusten Daten liefert, die die KI zum Trainieren braucht. Dieses Verfahren wird die Informationsextraktion erheblich beschleunigen – und die versteckten Daten schneller und trotzdem zuverlässig aufspüren. Die extrahierten Daten werden von dem Projektpartner TIB dann in einem im Projekt erstellten Knowledge Graph zur Verfügung gestellt.
Vom Genom zu phänotypischen Beschreibungen
Phänotypische Daten sind also zunächst meist unstrukturiert, anspruchsvoll und nur schwer zu bestimmen. Im Gegensatz dazu sind Genomsequenzen für eine große Anzahl von Bakterienarten immer häufiger verfügbar. Das bietet die Möglichkeit, aus Genomsequenzen phänotypische Informationen abzuleiten. Frühere Projekte haben diese Möglichkeiten bereits genutzt[1] [2]. Es hat sich dabei gezeigt, dass trotz einer begrenzten Anzahl an Trainingsdaten von weniger als 500 Bakterienstämmen die Vorhersage verschiedener Merkmale mit einer Genauigkeit von bis zu 99 % möglich war. DiASPora erweitert nun diese Ansätze, indem es auf weitaus größere Mengen an manuell kuratierten phänotypischen Daten aus BacDive zurückgreift. Inzwischen sind die Genome von fast 10.000 Stämmen sequenziert – ein Vielfaches der Daten, die für die vorherigen Projekte zur Verfügung standen.
Ziel ist es nun, die phänotypischen Daten dieser Stämme durch einen KI-gesteuerten Ansatz anzureichern. Die ersten Ergebnisse zeigten eine Genauigkeit zwischen 78 und 97 %. Der Ansatz bietet also ein großes Potenzial. Die Verfügbarkeit und Verteilung der Daten begrenzen ihn jedoch auch. Das Text Mining ist daher ein wichtiger Schritt, um die Verfügbarkeit zu verbessern.
Mit KI zur Vorhersage von Kultivierungsbedingungen für Bakterien
Was fängt man nun mit den Daten an? Die erweiterte Datenbasis in BacDive dient zum Beispiel der Vorhersage von Kultivierungsbedingungen für bisher nicht kultivierte Bakterien. Denn nur Bakterien, die sich in einer Kultur vermehrt haben, können auch weiter erforscht werden, was für medizinische und biotechnologische Anwendungen unabdingbar ist. Dafür kombiniert dieser Teil des Projektes, der vornehmlich durch das DSMZ umgesetzt wird, Datensätze aus einer Medienliste, die zuvor angewandten KI-Techniken und die vorhergesagten phänotypischen Daten und überführt sie in die neuentwickelte Datenbank BacMedia[3]. Diese Nährboden-Datenbank ist frei verfügbar und bietet Anleitungen zur Kultivierung von über 40.000 Mikroorganismenstämmen. Die Datenbank wird im Projekt unter anderem durch Praxistests noch weiterentwickelt.
Ausblick
DiASPora hat sich zum Ziel gesetzt, die Biodiversitätsinformationen über schlecht untersuchte Organismen zu verbessern – mit unterschiedlichen Ansätzen. Text Mining ermöglicht die Extraktion von verstecktem Wissen aus veröffentlichten Quellen, während KI-gestützte Vorhersagen von phänotypischen Merkmalen dieses Wissen extrapolieren. Die Transformation aller Daten in einen maschineninterpretierbaren Wissensgraphen wird innovative Suchoptionen für die Entdeckung neuer wissenschaftlicher Erkenntnisse und bis dato verborgener Datenbeziehungen ermöglichen. Und all diese Daten werden die BacDive-Datenbank erweitern und somit für die mikrobielle Forschungsgemeinschaft gemäß den FAIR-Grundsätzen zugänglich sein. Das DiASPora-Projekt wird so mit dem enormen Wissenszuwachs das gesamte Verständnis für die schlecht untersuchte Mehrheit der Bakterienarten in der Forschung verbessern.
Mehr Infos zu DiASPora – Digital Approaches for the Synthesis of Poorly Accessible Biodiversity Information
Dieser Beitrag erscheint parallel im ZB MED-Jahresbericht 2022.
DOI (Digitalaugabe): https://doi.org/10.48664/ksc2-pw24
[1] Weimann A et al. 2016 mSystems; 27;1(6):e00101-16. DOI: 10.1128/mSystems.00101-16.
[2] Feldbauer R et al. 2015 BMC Bioinformatics. 16 Suppl 14(Suppl 14):S1. DOI: 10.1186/1471-2105-16-S14-S1.
[3] bacmedia.dsmz.de






